联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
DeepMind公司开发的AlphaFold2人工智能系统,基于氨基酸序列,精确预测了蛋白质的3D结构。它的准确性与使用冷冻电子显微镜(CryoEM)、核磁共振或 X 射线晶体学等实验技术解析的3D结构相媲美。这一突破被誉为“变革生命科学和生物医学”的突破。
昨日,DeepMind公司在《自然》杂志上发表论文,公开了进一步优化的AlphaFold2人工智能系统的源代码并且详细描述了它的设计框架和训练方法。同日,华盛顿大学(University of Washington)蛋白设计研究所David Baker教授课题组在《科学》杂志上发表论文,公布了其开源人工智能系统RoseTTAFold的研究结果。《自然》上发表的评论指出,这些论文和人工智能系统资源的发布,不但让基于DeepMind开发的AI系统预测蛋白质结构的技术能够为广大科学家和研究人员使用,而且有望进一步激发这一领域的进展。

蛋白质对生命来说不可或缺,它们支持生物体的几乎所有功能。这些复杂的大分子由氨基酸链构成,而蛋白质的功能很大程度上决定于它的3D结构。生物医学领域的众多挑战,包括开发治疗疾病的创新疗法,依赖于对蛋白质结构和功能的理解。
在过去的五十年中,科学家们已经能够利用冷冻电子显微镜、核磁共振或 X 射线晶体学等实验手段在实验室中确定蛋白质的形状,但每种方法都依赖于大量的试错,耗时耗力,可能需要花上好几年时间。1972年,诺贝尔化学奖得主Christian Anfinsen博士表示,理论上,蛋白质的氨基酸序列应该能够完全决定它的3D结构。这一假说激发了50年来基于氨基酸序列,通过计算方法预测蛋白质3D结构的探索。
在2018年,DeepMind开发的AlphaFold人工智能系统***在国际蛋白质结构预测竞赛(CASP)上亮相。而在去年的CASP上,DeepMind的AlphaFold2系统表现惊艳,在接受检验的近100个蛋白靶点中,AlphaFold2对三分之二的蛋白靶点给出的预测结构与实验手段获得的结构相差无几。有些情况下,已经无法区分两者之间的区别是由于AlphaFold2的预测出现错误,还是实验手段产生的假象。
DeepMind发布的更新版AlphaFold2在去年的系统基础上进行了优化,在解析蛋白结构的速度上有了进一步的提高。主要开发人员John Jumper博士说,这一系统的处理速度快了大约16倍。根据蛋白的大小,它可以在几分钟到几小时内生成准确的蛋白结构。

AlphaFold2系统去年在CASP上的出色表现促进了蛋白质3D结构预测领域其它团队的进步。华盛顿大学蛋白设计研究所David Baker教授的团队从AlphaFold2的设计思路中获得启发,构建了名为RoseTTAFold的软件系统。它的神经网络能够同时考虑蛋白序列的模式,蛋白中不同氨基酸之间的相互作用,以及蛋白质可能出现的3D结构。在这个系统中,一维、二维和三维的信息能够相互交流,让神经网络综合所有信息,决定蛋白质的化学组成部分和它折叠产生的结构之间的关系。
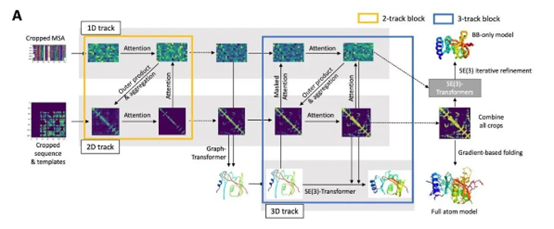
研究人员表示,RoseTTAFold系统在解析蛋白质3D结构方面的表现,与AlphaFold2的水平几乎相当,在有些蛋白上甚至优于AlphaFold2。
在论文中,研究人员指出,这一工具还可以用于预测由两个或者多个蛋白构成的复合体的构象。这让研究人员可以直接从蛋白序列,推测出不同蛋白相互结合的结构模型。在论文中,研究人员利用IL-12和IL-12受体(IL-12R)的序列预测的IL-12/IL-12R复合体结构与此前用冷冻电子显微镜解析的结构非常类似。
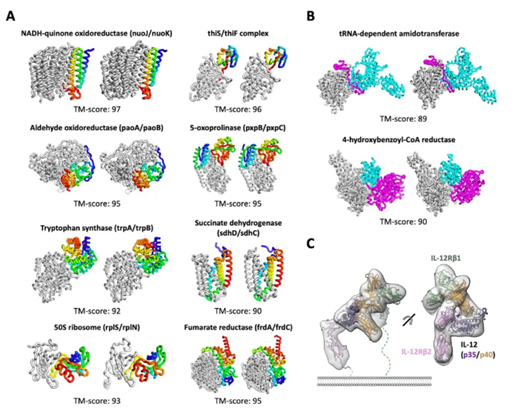
▲RoseTTAFold系统能够基于多个蛋白序列,预测复合体的结构
Baker教授的团队已经将RoseTTAFold软件工具上传到GitHub网站上,7月份以来,它已经被世界上超过140个不同研究团队下载。
同时,该团队也搭建了服务器,让研究人员可以输入蛋白质序列,然后获得预测的蛋白结构。服务器在上个月启动后,已经帮助解析了大约500名用户递交的超过5000个蛋白结构。“我很高兴科学界已经在利用RoseTTAFold服务器来解决重要的生物学问题。” David Baker教授说。
“我们希望这一新工具将继续为整个研发团体造福。”论文的***作者,Baker教授课题组的博士后Minkyung Baek说。
《自然》发表的新闻评论指出,随着RoseTTAFold和AlphaFold2源代码的公布,研究人员可以在两者的基础上继续前进,有望对人工智能系统做出进一步改进,攻克目前人工智能系统尚且无法确定构象的蛋白,以及使用这些软件设计全新的蛋白。