联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
《自然》本周上线的一篇论文中,由中国科学技术大学刘海燕教授、陈泉副教授领衔的研究团队展示了蛋白质从头设计的一条全新路线,尤其是为人工设计蛋白质的一大难题——如何充分地探索蛋白质主链结构空间,提供了一个系统性的解决方法。
《自然》杂志的审稿人对于这项研究成果给予了很高的评价,认为新方法“刻画了更传统的统计方法无法企及的***特征,具有足够的新颖性和实用性”。

蛋白质被认为是生命的基础,生命功能的主要执行者,它们的结构与功能由氨基酸序列决定。天然蛋白质的氨基酸序列经过长期自然进化,能够让蛋白质形成稳定的三维结构。然而,天然蛋白的结构功能还无法充分满足应用需求,于是人们试图通过设计氨基酸序列,创建出自然界中从未见过的蛋白质,使其具有特定的功能,比如可以对抗癌细胞或是对抗新兴的病毒。
研究团队介绍,在人工设计蛋白质时,目前国际上报道的主要方法是使用天然结构片段作为构建模块来拼接产生人工结构。但这类方法有其局限性,包括设计结果单一、对主链结构细节过于敏感,从而***了设计主链结构的多样性和可变性。
刘海燕教授、陈泉副教授团队十余年来致力于发展数据驱动的蛋白质设计方法,以期发现新颖的、“高可设计性”主链结构。经过长期不懈努力,他们突破了只能用天然片段来拼接产生新主链结构的***,设计出不同于已知天然蛋白的新颖结构。
在此次发表的工作中,研究人员建立了给定主链结构设计氨基酸序列的ABACUS模型,进而发展了能在氨基酸序列待定时从头设计全新主链结构的SCUBA(即Side Chain-Unknown Backbone Arrangement)模型。
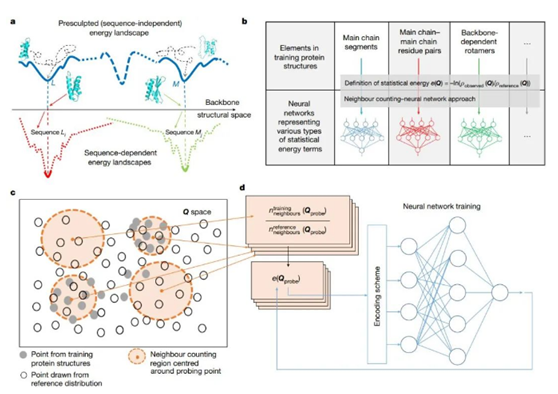
▲用SCUBA模型进行蛋白质设计的原理:(a)SCUBA主链能量面上的极小对应了蛋白质的可设计主链结构,即特定氨基酸序列下的***低自由能结构;(b)SCUBA中用神经网络表示的统计能量项;(c)和(d)用近邻计数(NC)-神经网络(NN)方法从蛋白质结构原始数据中学习解析能量函数的方法框架。
该统计模型的一大特点是采用神经网络形式的能量项。具体来说,基于核密度估计(或近邻计数,NC)和神经网络拟合(NN)方法,从原始结构数据中得到神经网络形式的解析能量函数,能够高保真地反应实际蛋白质结构中不同结构变量间的高维相关关系,在不确定序列的前提下,连续、广泛地搜索主链结构空间,自动产生“高可设计性”主链。
这种计算方法在此次研究中还得到了实验验证。团队报道了9种从头设计的蛋白质分子的X射线晶体结构(如下图所示),它们的实际结构与设计模型一致,其中5种蛋白质具有天然蛋白质中尚未观察到的新型拓扑结构。
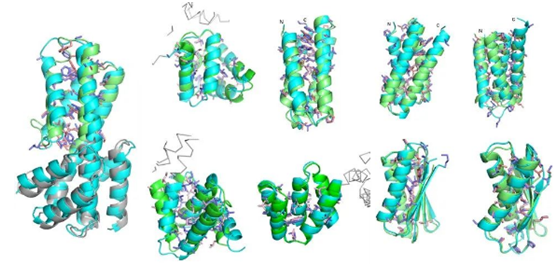
▲从头设计蛋白的高分辨晶体结构(天蓝色)与设计模型(绿色)比较
中国科学技术大学的新闻稿指出,这一工作在蛋白质设计的前沿科技领域实现了关键核心技术的原始创新,为工业酶、生物材料、生物医药蛋白等功能蛋白的设计奠定了坚实的基础。
生物磁珠对细胞筛选的方法已日渐成熟,原理是将包被一抗的磁珠与细胞表面对应的分子特异性结合,或者将包被二抗的磁珠与已经与细胞表面分子特异性结合的一抗结合。磁珠携带与之结合的细胞吸附与分离柱或试管上,实现阳性细胞或阴性细胞的分离。洛阳吉恩特生物自主研发生产了各类生物磁珠,可以实现稳定的实验结果。