联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
WES • 单基因病
全外显子组测序(Whole Exome Sequencing, WES)是指利用序列捕获技术对基因组外显子区域捕获并富集后进行高通量测序的基因组分析方法。对外显子区域的***分析有助于检测出和疾病相关的编码区突变,从而研究DNA变异对正常生理机制与疾病发病机制的影响。
单基因遗传病是指受一对等位基因影响的疾病,主要由遗传因素决定,遵循孟德尔遗传定律。目前全球已知明确致病机制的单基因疾病约7000种,并且每年以10-50种的速度递增,已经对人类健康构成了较大的威胁。随着二代测序技术的兴起,特别是外显子测序技术的出现,可以用较低的成本准确检测到蛋白质编码区的常见及罕见变异,极大地促进了疾病基因的识别。
在单基因遗传病的致病基因研究中,根据样本情况不同,可分为家系样本和散发样本研究,其中家系连锁分析法是***有效、准确的方法之一。
应用实例

这是2018年6月8日发表在Science上的一篇关于全外显子测序在单基因遗传病中的应用文章。
背景介绍
心血管疾病(CVD)是世界范围内的一类重要的致死疾病,其中血液中高浓度的低密度脂蛋白胆固醇(low-density lipoprotein cholesterol, LDL-C)是它的一个主要危险因素之一。LDL-C如果浓度过高,会沉积在动脉壁中,形成斑块,造成血管堵塞,从而引发心血管疾病。
LDL-C的浓度是一个受环境和遗传因素共同影响的复杂性状,其中大约40-50%的LDL-C的表型变异是由遗传因素导致的。已有大规模分析显示,只有2.5%的高浓度LDL-C患者具有已知的家族性遗传变异。此外GWAS研究发现,在所有的LDL-C相关突变中约10-20%的突变是共同的SNVs。这些已有结果说明,影响LDL-C的遗传因素尚未被完全揭示,且人群中自发基因变异差别巨大,因此对于不同人群的LDL-C遗传水平的研究具有重要意义。
材料选择
该文章选用的是家系样本。在中国西部心血管疾病风险调查中发现一个中国哈萨克族家系,命名为Family 1,该家系成员表现出家族聚集的低水平LDL-C。该家系的特点是:
1. 哈萨克族是中国西部的主要族群之一,是突厥和中世纪蒙古人的后代,在其基因组中显示出和汉族有明显的SNP差异;
2. 该民族独立生活,通常与自己的族内个体结婚;
3. 目前该民族还没有进行过脂质相关基因的研究。
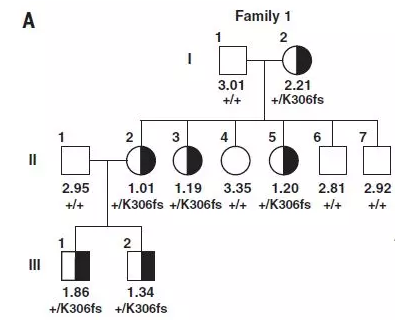
图1 低水平LDL-C的中国哈萨克族家系图
注:+/+正常浓度的LDL-C个体(2.7~3.36 mmol/L);+ / K306fs较低浓度的LDL-C个体(0.29~2.42 mmol/L)
研究思路
1. 样本选择思路
本研究对Family 1家系的case I:2(LDL-C低浓度个体)、case II:2(LDL-C低浓度个体)、case III:1(LDL-C低浓度个体)、case II:4(LDL-C正常浓度个体)四个个体进行全外显子测序,使用Illumina HiSeq 2000平台,PE100模式测序。在每一世代中选择了一个LDL-C低水平的个体,同时在II代中选择了一个LDL-C正常水平的女性个体作为对照,过滤无意义突变。文章在家系样本选择方面可以满足家系分析的需要。
2. 全外显子数据分析过滤思路
对家系样本进行全外显子测序之后,为了准确识别低水平LDL-C的突变位点,作者对全外显子组测序数据突变位点进行了多次过滤,数据分析过程如下:
***阶段:全外显子测序数据进行数据质量评估→突变寻找→突变注释流程,四个样本中各自检测到7000—8000多不等的突变位点;
第二阶段:突变位点识别过滤,对四个家系样本检测到的所有突变位点进行过滤
Filter 1:过滤掉不存在于LDL-C低水平个体中的突变位点、同时存在于LDL-C低水平个体和正常水平个体中的突变位点,剩余3333个突变位点;
Filter 2:过滤掉同义突变和内含子上的突变,剩余555个突变位点;
Filter 3:过滤掉dbSNP数据库中的已知突变、1000Genomes数据库中MAF>0.5%的突变、ESP6500和ExAC数据库中MAF>0.1%的突变,剩余24个突变位点;
Filter 4:过滤掉SIFT分值高的突变位点、POLYPhen V2和Mutation Taster分值低的突变位点,剩余7个突变位点;
Filter 5:通过一代测序验证,***终得到一个突变位点,即LIMA1基因上的一个移码缺失突变LIMA1-K306fs。
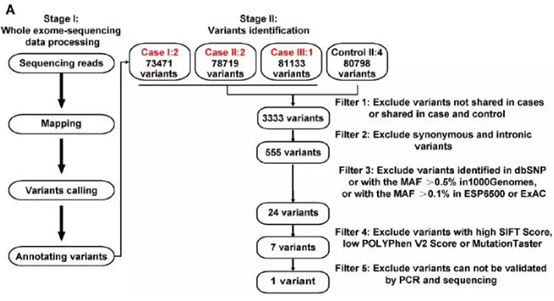
图2 全外显子组测序数据处理及突变鉴定示意图
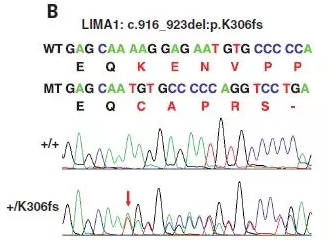
图3 突变位点的一代测序数据
3. 突变位点验证实验
经全外显子测序以及一代测序验证***终发现了一个未知突变LIMA1-K306fs。为了进一步验证突变结果的准确性,本研究继续对510个正常LDL-C浓度的中国哈萨克族个体和509个低LDL-C浓度的中国哈萨克族个体进行了LIMA1基因编码区的序列测定,发现正常个体中没有K306fs突变,低LDL-C浓度的个体中检测到K306fs杂合子。此外,在Dallas Heart 和 Biobank 共9400人的研究中都没有发现K306fs突变,说明该突变是不同人群中的一种罕见突变。对Family 1的9个成员进行全基因组连锁分析,发现连锁基因LIMA1位于12号染色体上,该突变发生在LIMA1基因第七个外显子上,产生一个提前终止子,造成LIMA1蛋白翻译提前终止,造成其分子量减少了60%。
人群验证实验:对1000多个哈萨克族个体全基因组的LIMA1基因片段进行靶向测序,发现另外3个家系的LIMA1基因中含有L25I突变(LIMA1-L25I (Leu→Ile))。在这三个家系中LIMA1-L25I突变与低LDL-C水平显著相关。L25I携带者(+/L25I)的血浆TC和LDL-C浓度较野生型显著降低,同时Ca:L也较低。当在细胞中表达时,L25I突变并不影响LIMA1基因表达RNA的稳定性或翻译效率,而是增加了蛋白降解速率。+/L25I携带者的LDL-C水平不如+/K306fs携带者低,这表明L25I可能通过破坏蛋白质的稳定性部分损害LIMA1的功能。
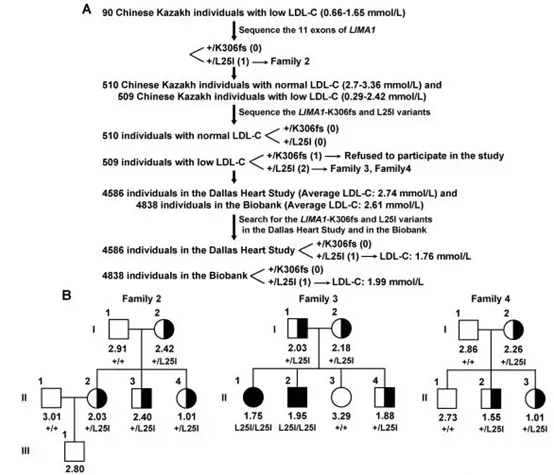
图4 A. 另外3个中国哈萨克族低水平LDL-C家族LIMA1-L25I突变鉴定示意图
B. 低水平LDL-C的哈萨克族2、3、4家系图
4. LIMA1作用机制研究
a )LIMA1的表达模式探究:利用小鼠模型进行免疫印迹实验、qPCR,观察小鼠LIMA1和NPC1L1(胆固醇吸收的关键跨膜蛋白)的表达情况。发现LIMA1在小鼠的小肠中高度表达,在肝脏中适度表达,在心脏、脾脏、肺、脑和胰腺中检测不到,并将其定位在小肠上皮刷状缘膜上。小肠特异性Lima1缺陷小鼠(I-Lima1 - / - )实验,证实LIMA1的缺陷不影响NPC1L1的表达水平。
b ) LIMA1功能验证:构建lima1缺陷小鼠,通过免疫荧光染色实验,研究LIMA1突变后小鼠对于饮食胆固醇的吸收,以及评估LIMA1在饮食引起的高胆固醇血症中的作用。通过检测小鼠体内目标器官中的胆固醇含量,发现LIMA1缺陷小鼠降低了膳食胆固醇的吸收,并对饮食诱导的高胆固醇血症具有抵抗力。
c ) 进行药理学实验:利用Ezetimibe来抑制NPC1L1的活性,以确定LIMA1是否在NPC1L1上游对胆固醇吸收产生影响。证明LIMA1作用于NPC1L1的上游,影响胆固醇的吸收。
d ) 通过免疫沉淀结合质谱法检测LIMA1的结合蛋白,发现LIMA1蛋白能结合NPC1L1和myosin Vb蛋白。
e ) 小鼠小肠免疫荧光染色实验表明LIMA1、NPC1L1和myosin Vb蛋白在小肠绒毛上皮细胞刷状缘膜上存在共定位。
f ) 在细胞水平,通过siRNA干扰LIMA1和myosin Vb的表达,以及构建过表达LIMA1(1-306)与LIMA1竞争性连接NPC1L1,发现LIMA1蛋白参与NPC1L1蛋白从内吞循环体(ERC)向质膜(PM)的细胞内转运过程。细胞和分子水平研究表明LIMA1蛋白介导NPC1L1蛋白和myosin Vb蛋白相互作用,从而参与NPC1L1蛋白的细胞内转运。阻断LIMA1和NPC1L1的结合可以抑制NPC1L1介导的胆固醇吸收。