联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
中国科学院北京生命科学研究院研究员赵方庆团队在Nature Biotechnology上发表了题为Comprehensive profiling of circular RNAs with nanopore sequencing and CIRI-long的论文,研究内容为***测定环形RNA全长转录本的实验和计算方法。该研究主要是利用随机引物对环形RNA进行的滚环反转录扩增,而后应用纳米孔测序技术对环形RNA的全长序列进行直接测序,并使用开发的CIRI-long算法识别长测序读段中的环形RNA序列并进行全长重构。实验结果表明,与传统的环形RNA二代测序技术相比,该方法将环形RNA检测灵敏度提升了20倍,并可实现对不同长度(<100bp-5kb)的环形RNA全长序列的无偏识别,大幅提升了环形转录本的重构能力,为其功能研究提供了重要的实验方法和计算工具。
环形RNA是一类在真核生物中广泛存在的具有特殊环状结构的RNA分子。研究表明,在生物体内,环形RNA主要通过其序列特征,发挥miRNA海绵、RBP海绵及翻译短肽等重要的生物学功能。因而环形RNA的全长序列确定是进行环形RNA功能研究的重要基础。由于环形RNA的内部序列与线性mRNA分子高度相似,在数据中很难区分来自环形RNA和线性RNA分子的读段。研究方法对于环形RNA结构的识别能力主要被二代测序的读长所***,对于长度较长(>500bp)的环形RNA分子,仍缺少有效的全长重构手段。
针对这一问题,研究团队构建并优化环形RNA建库流程,使用随机引物对环形RNA进行滚环反转录扩增,结合片段长度筛选(~1kb),针对长度更长的目标cDN***段进行富集,***后使用纳米孔测序技术,实现了对目标环形RNA的全长序列进行直接测定。同时,研究人员进一步开发了CIRI-long算法,识别纳米孔测序数据中的环形RNA结构,并使用偏序比对算法,校正纳米孔测序带来的测序错误。随后,CIRI-long基于基因注释和剪接信号信息,提供单一样本内和多样本间结果的整合与校正方法,实现了环形RNA的准确识别和全长重构。
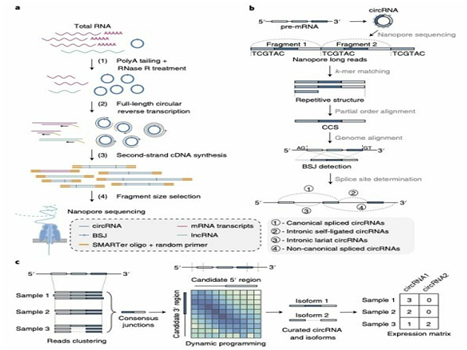
为了评估CIRI-long方法的准确性和效率,研究人员利用模拟数据和多次实验重复,综合评估CIRI-long结果,发现该方法具有较高的灵敏度,且与实验结果保持了高度的一致性。同时,结合二代测序结果及公共数据库的***分析,表明CIRI-long可有效地对高表达环形RNA进行识别,且与二代测序方法相比,CIRI-long对环形RNA的检测效率有着近20倍的提升,对表达丰度较低的环形RNA有着更好的识别效果,同时可以识别到长度更长的环形RNA分子,大幅提升了环形RNA全长的检测能力。
利用该方法,研究进一步发现了一类由内含子自连形成的新型环形RNA分子(Intronic self-ligated circRNA)。这类环形RNA具有特殊的剪接位点内侧的GT-AG信号和较高的两侧序列保守性,在之前工作中缺乏普遍研究。此外,研究人员在小鼠不同组织中,对内含子自连型环形RNA的表达模式开展了进一步探究,发现Tpm1基因可以产生一个长度为767bp的内含子自连型环形RNA——circTpm1。与Tpm1基因的其他内含子相比,该分子成环区域具有相对较高的保守性,且与线性mRNA在组织间具有不同的表达模式,说明circTpm1并非其母本基因转录的副产物,可能具有一定的生物学功能。
综上,研究人员开发了基于纳米孔测序技术的环形RNA全长识别流程CIRI-long,通过结合滚环反转录扩增和纳米孔长读长测序技术,可直接测定环形RNA的全长序列,实现了对环形RNA的高灵敏度检测和内部结构重构。