联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
蛋白质存在于每一个细胞中,参与到基因表达、细胞发育和代谢等各种过程。它们由氨基酸搭建成线性的分子,并且会自发折叠成3D的形状。参与构建的氨基酸可以从几十个到几千个不等。 尽管氨基酸和蛋白质都很常见,但不代表人类真的能参透其中的机制。原因在于根据不同的氨基酸和序列,蛋白质能折叠成的构型数量是一个天文数字。 为了破解其中的秘密,生物学界曾于1994年开始举办国际蛋白质结构预测竞赛(CASP)来吸引全世界***前沿的实验室挑战预测蛋白折叠。CASP使用称为GDT的评分系统来评估预测蛋白结构的精确性。这个评分从0到100,如果评分达到90分以上,可以认为预测的结构与实验手段获得的结构相当。 2018年,DeepMind开发的***代AlphaFold***参加CASP就拔得了头筹,而在2020年的第14届CASP中,AlphaFold系统对所有蛋白靶点3D结构预测的中位GDT评分为92.4分。即便是针对***难解析的蛋白靶点,AlphaFold的中位GDT评分也达到了87.0分。在接受检验的近100个蛋白靶点中,AlphaFold对三分之二的蛋白靶点给出的预测结构与实验手段获得的结构相差无几。
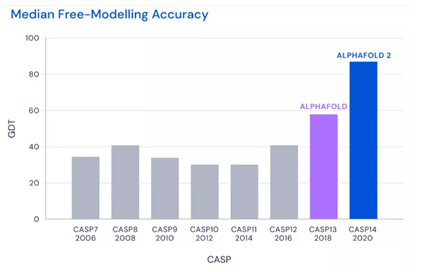
▲2006-2020年CASP比赛中***佳蛋白折叠预测系统的评分表现
今年7月,华盛顿大学David Baker教授的实验室在《科学》上发表了不同于AlphaFold策略的RoseTTAFold算法,它同样能够在10分钟根据氨基酸序列预测出蛋白结构。而就在上个月,两种AI更是联手预测了真核生物蛋白质复合体的三维结构,实现了从单体到复合体的突破。
但我们要知道的是,AI能带来的惊喜总是没有上限的。这一次,《自然》的新研究展示了AI的强大想象能力,它不仅能预测结构,甚至能自己幻想出新的蛋白结构,再次震惊了学界!

新研究中,研究者不再给AI(trRosetta)一些已知蛋白结构的氨基酸序列进行预测,而是向它们提供一些随机的序列,并且会向其中引入一些突变,直到AI的神经网络确定能够将这些序列折叠成稳定的结构为止。“我们不会在任何时候引导AI朝特定方向前进,一切都依靠它们的想象,”在Baker实验任职的Ivan Anishchenko博士表示。 这一点有些类似Google DeepDream的工作原理,DeepDream的网络会通过大量的训练识别面部和其他模式的照片,然后将这些“知识”逆向使用,通过模糊的照片推测出清晰的模式。而新研究同样如此,trRosetta的神经网络已经在许多预测中获取了许多有关蛋白序列和结构的信息,现在便是时候让它逆向推测了,研究者给它一个随机的未知(模糊)序列,直到它能想象出稳定的结构(清晰)。
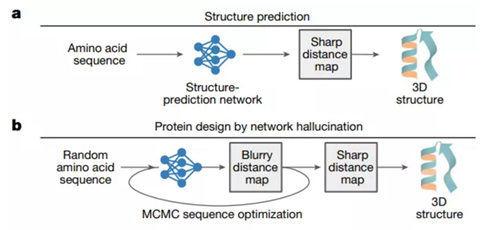
▲新研究设计的AI“想象”策略
研究中提供的都是包含100个氨基酸的随机序列,这些氨基酸之间的距离分布和特点要比天然蛋白的更加模糊。为了让AI想象出的蛋白差异化更明显,研究者还会在随机的位置替换掉一些氨基酸,改变残基对分布序列。 在大量训练和学习之后,trRosetta利用随机的序列产生了2000种全新的蛋白结构,这些结构与天然蛋白相似性非常低。此外,这些想象的结构非常多样化,彼此之间有很大差别。这些蛋白都含有常规的α螺旋和β折叠,但并没有天然蛋白会有的环状和其他结构。
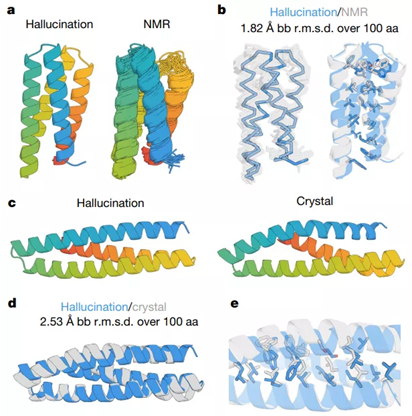
▲想象出来的蛋白结构和实际形成的相差无几
此外,Baker实验室与华盛顿大学合作,他们获得了129个“想象”蛋白的合成基因,然后通过基因工程在大肠杆菌中获得了这些蛋白。通过结合核磁共振和X射线晶体成像技术,研究者确定AI想象的蛋白结构和实际形成的结构几乎是一样的。“它们想象出的蛋白结构准确率非常高,即使这些序列在天然蛋白没有相近的序列,” Anishchenko博士表示。 当AI有了想象能力,医学的未来发展将变得完全难以预料。当研究者有工具任意地设计想要的蛋白时,任何以蛋白为基础的药物、酶都能随心所欲的获取。